干擾素基因刺激蛋白
干擾素基因刺激蛋白( STING ),也稱為跨膜蛋白 173 ( TMEM173 ) 和MPYS / MITA / ERIS ,是人類中由 STING1基因編碼的蛋白質。[6]
STING 在先天免疫中發揮着重要作用。當細胞被病毒、分枝桿菌和細胞內寄生蟲等細胞內病原體感染時,STING 會誘導I型干擾素產生。[7] I 型干擾素由 STING 介導,通過與分泌干擾素的同一細胞(自分泌信號)和附近的細胞(旁分泌信號)結合,保護受感染的細胞和附近的細胞免受局部感染。 )因此,它在控制諾如病毒感染等方面發揮着重要作用。[8]
STING 既可作為直接胞質 DNA 傳感器(CDS),又可通過不同的分子機製作為I 型乾擾素信號轉導中的銜接蛋白。它已被證明可以通過TBK1激活下游轉錄因子STAT6和IRF3 ,這些因子負責抗病毒反應和針對細胞內病原體的先天免疫反應。[9]
結構
[編輯]
人類 STING 的氨基酸 1-379 包括 4 個跨膜區(TM) 和一個C 端結構域。 C 末端結構域(CTD:氨基酸 138-379)包含二聚結構域(DD)和羧基末端尾部(CTT:氨基酸 340-379)。[9]
STING 在細胞中形成對稱二聚體。 STING 二聚體類似於蝴蝶,兩個原聚體之間有一個深裂縫。每個 STING 原聚體的疏水殘基在界面處彼此之間形成疏水相互作用。[9]
表達
[編輯]STING在外周淋巴組織的造血細胞中表達,包括T淋巴細胞、 NK細胞、骨髓細胞和單核細胞。研究還表明,STING 在肺、卵巢、心臟、平滑肌、視網膜、骨髓和陰道中高表達。[10][11]
定位
[編輯]STING 的亞細胞定位已被闡明為內質網蛋白。此外,STING 很可能與線粒體相關內質網膜 (MAM)(線粒體和內質網之間的界面)緊密相連。[12]在細胞內感染期間,STING 能夠從內質網重新定位到可能參與外囊介導的運輸的核周囊泡。[12]在雙鏈 DNA 刺激後,STING 還被證明與自噬蛋白、微管相關蛋白 1 輕鏈 3 (LC3)和自噬相關蛋白 9A共定位,表明它存在於自噬體中。[13]
功能
[編輯]STING 介導I 型乾擾素的產生,以響應細胞內 DNA 和多種細胞內病原體,包括病毒、細胞內細菌和細胞內寄生蟲。[14]感染後,受感染細胞的 STING 可以感知細胞內病原體核酸的存在,然後誘導干擾素 β和 10 多種形式的干擾素α產生。受感染細胞產生的I型乾擾素可以發現並結合附近細胞的干擾素α/β受體,從而保護細胞免受局部感染。
抗病毒免疫力
[編輯]STING 可引發強大的I 型乾擾素免疫力,抵抗病毒感染。病毒進入後,病毒核酸存在於受感染細胞的細胞質中。多種DNA傳感器,如DAI 、 RNA聚合酶III 、 IFI16 、 DDX41和cGAS ,可以檢測外源核酸。識別病毒 DNA 後,DNA 傳感器通過激活 STING 介導的干擾素反應來啟動下游信號通路。[15]
腺病毒、單純疱疹病毒、HSV-1 和 HSV-2 以及負鏈 RNA 病毒、水泡性口炎病毒(VSV) 已被證明能夠激活 STING 依賴性先天免疫反應。[16]
由於缺乏成功的 I 型干擾素反應,小鼠 STING 缺陷導致對 HSV-1 感染的致命易感性。[17]
Serine-358 的點突變抑制了蝙蝠中 STING-IFN 的激活,並被認為賦予蝙蝠作為儲存宿主的能力。[18]
對抗細胞內細菌
[編輯]細胞內細菌(李斯特菌)已被證明可以通過 STING 刺激宿主免疫反應。[19] STING 可能在MCP-1和CCL7趨化因子的產生中發揮重要作用。在李斯特菌感染期間,STING 缺陷單核細胞在向肝臟遷移方面存在本質缺陷。通過這種方式,STING 通過調節單核細胞遷移來保護宿主免受感染。 STING 的激活可能是由細胞內細菌分泌的環二-AMP介導的。[19][20]
其他
[編輯]STING 可能是針對傳染性生物體的保護性免疫的重要分子。例如,不能表達STING的動物更容易受到VSV 、 HSV-1和李斯特菌的感染,這表明其與人類傳染病的潛在相關性。[21]
在宿主免疫中的作用
[編輯]儘管I 型乾擾素對於抵抗病毒絕對至關重要,但越來越多的文獻表明I 型乾擾素在 STING 介導的宿主免疫中的負面作用。惡性惡性瘧原蟲和伯氏瘧原蟲基因組中富含 AT 的莖環 DNA 基序以及結核分枝桿菌的細胞外 DNA 已被證明可以通過 STING 激活I 型乾擾素。[22][23] ESX1分泌系統介導的吞噬體膜的穿孔允許細胞外分枝桿菌DNA進入宿主細胞質DNA傳感器,從而誘導巨噬細胞產生I型干擾素。高I 型干擾素特徵導致結核分枝桿菌發病機制和長期感染。[23] STING-TBK1-IRF 介導的I 型乾擾素反應對於感染伯氏瘧原蟲的實驗動物中實驗性腦型瘧疾的發病機制至關重要。缺乏I 型乾擾素反應的實驗小鼠對實驗性腦型瘧疾有抵抗力。[22]
STING信號機制
[編輯]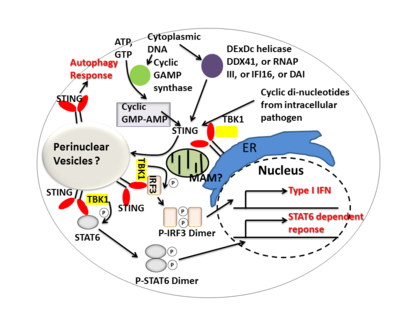
STING 通過充當直接 DNA 傳感器和信號轉接蛋白來介導I 型乾擾素免疫反應。激活後,STING 會刺激TBK1活性,使IRF3或STAT6磷酸化。磷酸化的IRF3和STAT6二聚化,然後進入細胞核刺激參與宿主免疫反應的基因表達,如IFNB 、 CCL2 、 CCL20等[9][24]。cGAS 與雙鏈 DNA 結合,激活其酶活性後將 ATP 及 GTP 轉化成第二信使 cGAMP,與內質網表面 STING 結合。STING 在結合後變化構 象並轉移至高爾基體結合 TANK 結合激酶 1(TANK binding kinase 1,TBK1)後 磷酸化干擾素調節因子(interferon regulatory factor,IRF)3,使其進入細胞核誘導干擾素表達。另外 STING 也可激活 IKK,磷酸化 IκB 家族促進 NF-κB 表達[25]。
一些報告表明 STING 與選擇性自噬的激活有關。[13]結核桿菌已被證明能產生激活 STING 的胞質 DNA 配體,導致細菌泛素化並隨後招募自噬相關蛋白,所有這些都是「選擇性」自噬靶向和針對結核桿菌的先天防禦所必需的。[26]
總之,STING 協調對感染的多種免疫反應,包括誘導干擾素和 STAT6 依賴性反應和選擇性自噬反應。[9]
作為細胞質 DNA 傳感器
[編輯]在細胞內病原體感染期間,在哺乳動物細胞的胞漿中檢測到由不同細菌種類產生的環狀二核苷酸-第二信使信號分子;這會導致TBK1 - IRF3的激活以及下游I 型乾擾素的產生。[9][27] STING 已被證明可以直接與環二 GMP結合,這種識別會導致細胞因子的產生,例如I 型乾擾素,這對於成功消除病原體至關重要。[28]胞質 cGAS 不具備核苷酸序列特異性,因此 STING 通路也 受自身 DNA 激活,細胞核泄漏或線粒體損傷時所釋放的 DNA 可能過度激活 STING 通路,引起炎症反應甚至自身免疫疾病。
作為信號適配器
[編輯]DDX41是解旋酶 DEXDc 家族的成員,在骨髓樹突細胞中識別細胞內 DNA 並通過與 STING 直接關聯介導先天免疫反應。[29]其他 DNA 傳感器 - DAI 、 RNA 聚合酶 III 、 IFI16也已被證明可以通過直接或間接相互作用激活 STING。[15]
環 GMP-AMP 合酶(cGAS) 屬於核苷酸轉移酶家族,能夠識別胞質 DNA 內容物,並通過產生第二信使環鳥苷單磷酸 - 腺苷單磷酸(環 GMP-AMP 或 cGAMP)來誘導 STING 依賴性干擾素反應。環 GMP-AMP結合的 STING 被激活後,它會增強TBK1磷酸化IRF3和STAT6的活性,從而產生下游I 型干擾素反應。[30][31]
有人提出,細胞內鈣在 STING 通路的反應中發揮重要作用。[32]
相關
[編輯]- STING agonist——位於5號人類染色體的基因
參考
[編輯]- ^ 與干扰素基因刺激蛋白相關的疾病;在維基數據上查看/編輯參考.
- ^ 2.0 2.1 2.2 GRCh38: Ensembl release 89: ENSG00000184584、ENSG00000288243 - Ensembl, May 2017
- ^ 3.0 3.1 3.2 GRCm38: Ensembl release 89: ENSMUSG00000024349 - Ensembl, May 2017
- ^ Human PubMed Reference:. National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Mouse PubMed Reference:. National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ STING1 stimulator of interferon response cGAMP interactor 1 [ Homo sapiens (human) ]. [2023-07-03]. (原始內容存檔於2023-07-03).
- ^ Nakhaei P, Hiscott J, Lin R. STING-ing the antiviral pathway. Journal of Molecular Cell Biology. Jun 2010, 2 (3): 110–2. PMID 20022884. doi:10.1093/jmcb/mjp048
 .
.
- ^ NYu P, Miao Z, Li Y, Bansal R, Peppelenbosch MP, Pan Q. cGAS-STING effectively restricts murine norovirus infection but antagonizes the antiviral action of N-terminus of RIG-I in mouse macrophage. Gut Microbes. 2021, 13 (1): 1959839. ISSN 1949-0976. PMC 8344765
 . PMID 34347572. doi:10.1080/19490976.2021.1959839
. PMID 34347572. doi:10.1080/19490976.2021.1959839  .
.
- ^ 9.0 9.1 9.2 9.3 9.4 9.5 Burdette DL, Vance RE. STING and the innate immune response to nucleic acids in the cytosol. Nature Immunology. Jan 2013, 14 (1): 19–26. PMID 23238760. S2CID 7968532. doi:10.1038/ni.2491.
- ^ EST expression profile of TMEM173. biogps org. biogps.org. [2023-07-03]. (原始內容存檔於2011-08-20).
- ^ NCBI TMEM173 expression GEOprofile. NCBI. www.ncbi.nlm.nih.gov/geoprofiles. [2023-07-03]. (原始內容存檔於2023-07-03).
- ^ 12.0 12.1 Ishikawa H, Barber GN. STING is an endoplasmic reticulum adaptor that facilitates innate immune signalling. Nature. Oct 2008, 455 (7213): 674–8. Bibcode:2008Natur.455..674I. PMC 2804933
 . PMID 18724357. doi:10.1038/nature07317.
. PMID 18724357. doi:10.1038/nature07317.
- ^ 13.0 13.1 Saitoh T, Fujita N, Hayashi T, Takahara K, Satoh T, Lee H, Matsunaga K, Kageyama S, Omori H, Noda T, Yamamoto N, Kawai T, Ishii K, Takeuchi O, Yoshimori T, Akira S. Atg9a controls dsDNA-driven dynamic translocation of STING and the innate immune response. Proceedings of the National Academy of Sciences of the United States of America. Dec 2009, 106 (49): 20842–6. Bibcode:2009PNAS..10620842S. PMC 2791563
 . PMID 19926846. doi:10.1073/pnas.0911267106
. PMID 19926846. doi:10.1073/pnas.0911267106  .
.
- ^ Barber GN. Innate immune DNA sensing pathways: STING, AIMII and the regulation of interferon production and inflammatory responses. Current Opinion in Immunology. Feb 2011, 23 (1): 10–20. PMC 3881186
 . PMID 21239155. doi:10.1016/j.coi.2010.12.015.
. PMID 21239155. doi:10.1016/j.coi.2010.12.015.
- ^ 15.0 15.1 Keating SE, Baran M, Bowie AG. Cytosolic DNA sensors regulating type I interferon induction (PDF). Trends in Immunology. Dec 2011, 32 (12): 574–81. PMID 21940216. doi:10.1016/j.it.2011.08.004. hdl:2262/68041
 .
.
- ^ 引用錯誤:沒有為名為
pmid21239155的參考文獻提供內容 - ^ Ma Z, Damania B. The cGAS-STING Defense Pathway and Its Counteraction by Viruses. Cell Host & Microbe. February 2016, 19 (2): 150–8. PMC 4755325
 . PMID 26867174. doi:10.1016/j.chom.2016.01.010.
. PMID 26867174. doi:10.1016/j.chom.2016.01.010.
- ^ Xie J, Li Y, Shen X, Got G, Zhu Y, Cui J, Wang L, Shi Z, Zhou P. Dampened STING-Dependent Interferon Activation in Bats. Cell Host & Microbe. March 2018, 23 (3): 297–301.e4. PMC 7104992
 . PMID 29478775. doi:10.1016/j.chom.2018.01.006
. PMID 29478775. doi:10.1016/j.chom.2018.01.006  .
.
- ^ 19.0 19.1 Jin L, Getahun A, Knowles HM, Mogan J, Akerlund LJ, Packard TA, Perraud AL, Cambier JC. STING/MPYS mediates host defense against Listeria monocytogenes infection by regulating Ly6C(hi) monocyte migration. Journal of Immunology. Mar 2013, 190 (6): 2835–43. PMC 3593745
 . PMID 23378430. doi:10.4049/jimmunol.1201788.
. PMID 23378430. doi:10.4049/jimmunol.1201788.
- ^ Woodward JJ, Iavarone AT, Portnoy DA. c-di-AMP secreted by intracellular Listeria monocytogenes activates a host type I interferon response. Science. Jun 2010, 328 (5986): 1703–5. Bibcode:2010Sci...328.1703W. PMC 3156580
 . PMID 20508090. doi:10.1126/science.1189801.
. PMID 20508090. doi:10.1126/science.1189801.
- ^ Ishikawa H, Ma Z, Barber GN. STING regulates intracellular DNA-mediated, type I interferon-dependent innate immunity. Nature. Oct 2009, 461 (7265): 788–92. Bibcode:2009Natur.461..788I. PMC 4664154
 . PMID 19776740. doi:10.1038/nature08476.
. PMID 19776740. doi:10.1038/nature08476.
- ^ 22.0 22.1 Sharma S, DeOliveira RB, Kalantari P, Parroche P, Goutagny N, Jiang Z, Chan J, Bartholomeu DC, Lauw F, Hall JP, Barber GN, Gazzinelli RT, Fitzgerald KA, Golenbock DT. Innate immune recognition of an AT-rich stem-loop DNA motif in the Plasmodium falciparum genome. Immunity. Aug 2011, 35 (2): 194–207. PMC 3162998
 . PMID 21820332. doi:10.1016/j.immuni.2011.05.016.
. PMID 21820332. doi:10.1016/j.immuni.2011.05.016.
- ^ 23.0 23.1 Manzanillo PS, Shiloh MU, Portnoy DA, Cox JS. Mycobacterium tuberculosis activates the DNA-dependent cytosolic surveillance pathway within macrophages. Cell Host & Microbe. May 2012, 11 (5): 469–80. PMC 3662372
 . PMID 22607800. doi:10.1016/j.chom.2012.03.007.
. PMID 22607800. doi:10.1016/j.chom.2012.03.007.
- ^ Chen H, Sun H, You F, Sun W, Zhou X, Chen L, Yang J, Wang Y, Tang H, Guan Y, Xia W, Gu J, Ishikawa H, Gutman D, Barber G, Qin Z, Jiang Z. Activation of STAT6 by STING is critical for antiviral innate immunity. Cell. Oct 2011, 147 (2): 436–46. PMID 22000020. doi:10.1016/j.cell.2011.09.022
 .
.
- ^ Motwani, Mona; Pesiridis, Scott; Fitzgerald, Katherine A. DNA sensing by the cGAS–STING pathway in health and disease. Nature Reviews Genetics. 2019-07-29, 20 (11). ISSN 1471-0056. doi:10.1038/s41576-019-0151-1.
- ^ Watson RO, Manzanillo PS, Cox JS. Extracellular M. tuberculosis DNA targets bacteria for autophagy by activating the host DNA-sensing pathway. Cell. Aug 2012, 150 (4): 803–15. PMC 3708656
 . PMID 22901810. doi:10.1016/j.cell.2012.06.040.
. PMID 22901810. doi:10.1016/j.cell.2012.06.040.
- ^ McWhirter SM, Barbalat R, Monroe KM, Fontana MF, Hyodo M, Joncker NT, Ishii KJ, Akira S, Colonna M, Chen ZJ, Fitzgerald KA, Hayakawa Y, Vance RE. A host type I interferon response is induced by cytosolic sensing of the bacterial second messenger cyclic-di-GMP. The Journal of Experimental Medicine. Aug 2009, 206 (9): 1899–911 [2023-07-03]. PMC 2737161
 . PMID 19652017. doi:10.1084/jem.20082874. (原始內容存檔於2018-07-20).
. PMID 19652017. doi:10.1084/jem.20082874. (原始內容存檔於2018-07-20).
- ^ Burdette DL, Monroe KM, Sotelo-Troha K, Iwig JS, Eckert B, Hyodo M, Hayakawa Y, Vance RE. STING is a direct innate immune sensor of cyclic di-GMP. Nature. Oct 2011, 478 (7370): 515–8. Bibcode:2011Natur.478..515B. PMC 3203314
 . PMID 21947006. doi:10.1038/nature10429.
. PMID 21947006. doi:10.1038/nature10429.
- ^ Zhang Z, Yuan B, Bao M, Lu N, Kim T, Liu YJ. The helicase DDX41 senses intracellular DNA mediated by the adaptor STING in dendritic cells. Nature Immunology. Oct 2011, 12 (10): 959–65. PMC 3671854
 . PMID 21892174. doi:10.1038/ni.2091.
. PMID 21892174. doi:10.1038/ni.2091.
- ^ Wu J, Sun L, Chen X, Du F, Shi H, Chen C, Chen ZJ. Cyclic GMP-AMP is an endogenous second messenger in innate immune signaling by cytosolic DNA. Science. Feb 2013, 339 (6121): 826–30. Bibcode:2013Sci...339..826W. PMC 3855410
 . PMID 23258412. doi:10.1126/science.1229963.
. PMID 23258412. doi:10.1126/science.1229963.
- ^ Sun L, Wu J, Du F, Chen X, Chen ZJ. Cyclic GMP-AMP synthase is a cytosolic DNA sensor that activates the type I interferon pathway. Science. Feb 2013, 339 (6121): 786–91. Bibcode:2013Sci...339..786S. PMC 3863629
 . PMID 23258413. doi:10.1126/science.1232458.
. PMID 23258413. doi:10.1126/science.1232458.
- ^ Evidence for a role of calcium in STING signaling. 4 Jun 2017. bioRxiv 10.1101/145854
 .
.





